読み物:がん早期発見のためのツールを拡大するターゲットRNAシーケンス
ターゲットRNA-seq法を用いて、様々な種類のサンプルから長鎖非コードRNAを濃縮し、効率的にシーケンスする最近の研究を紹介します。

RNAシーケンス(RNA-seq)は、がんの早期発見に大きな可能性を秘めた重要な研究ツールですが、臨床サンプルではmRNAやrRNAの高い存在量によって、がんの早期発見に重要な鍵を握る可能性のある希少なRNAサブタイプ、特に長鎖非コードRNAは隠れてしまいます。今回、ゲント大学の研究チームが、ターゲットRNA-seqを用いてさまざまなサンプルからlncRNAを効率的に濃縮する方法を開発した研究について紹介し、最新の技術革新が高解像度RNA-seq実験においていかに重要な役割を果たすかを議論します。
一般的に、がんの治療は早期発見がカギとなり、早期に発見されるほど治療が成功しやすくなります。2018年の英国の報告によると、がんの55%がステージ1または2の段階で発見されています。しかし、がんの種類によって早期発見率は大きく異なり、精巣がんでは90%が早期発見される一方、膵臓がんではわずか22%でした(参考文献1)。
研究者たちは長い間、非侵襲的かつ組織へのダメージが少なく、がん細胞のわずかな集団を検出する感度を持つスクリーニング方法を模索してきました。この努力の中で、長鎖非コードRNA(lncRNA)と呼ばれる遺伝要素への関心が高まっています。
lncRNAは、200塩基以上の長さを持ち、タンパク質コード情報を翻訳しないRNA分子を含む多様なカテゴリに属します。現在、数千のlncRNA転写産物が確認されており、その中にはがんの発症や進行に重要な役割を果たすものもあると考えられています。がん関連のlncRNA転写産物は、患者の血液中にも存在することが確認されており、液体生検とRNA-seqを組み合わせることで、lncRNAの検出が有望な診断ツールになる可能性があります。
しかし、lncRNA転写産物については、どのような状況で発現するのかも含め、ほとんど解明されていません。その原因は、lncRNAのシーケンスと同定が非常に困難であるという技術的な制約にあります(参考文献2-4)。
幸いなことに、オリゴヌクレオチド合成とRNA-seqの進歩により、lncRNAの謎が少しずつ解明されつつあります。ここでは、この分野における最新の進展について紹介します。ベルギーのゲント大学の研究者たちは、高感度なRNA-seq手法を開発し、さまざまなサンプルからlncRNA転写産物を同定することに成功しました。この技術は、がんの早期発見に有望なツールとなる可能性を秘めています。
ターゲットRNA-seqとトータルRNA-seqの比較
lncRNAの配列と機能を研究することは、いくつかの課題を伴います。メッセンジャーRNA(mRNA)やリボソームRNA(rRNA)と比較して、lncRNAは非常に低レベルで発現するため(参考文献3,5)、通常のRNA-seqではmRNAやrRNAに隠れてlncRNAが検出されにくくなっています。
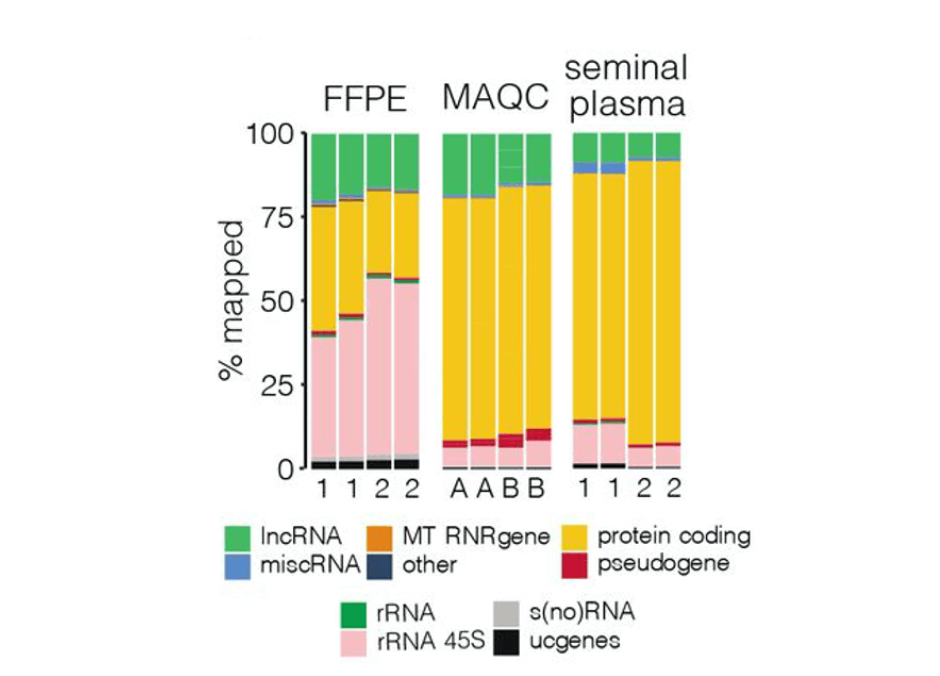
ヒトサンプルの大部分はrRNAおよびタンパク質コードRNAで構成されています。データによると、さまざまなサンプルタイプにおけるトータルRNAシーケンス(RNA-seq)でマッピングされたリードの割合が示されています。FFPEはホルマリン固定パラフィン包埋サンプル、MAQCは定量PCRヒト参照トータルRNAサンプルを指します。データはMorlionら(2021)の図1aから引用され、CC-by-4.0ライセンスの下で公開されています。
この課題を克服する一つの方法がターゲットRNA-seqです。ターゲットRNA-seqでは、広範な遺伝物質のプールをフィルタリングして特定のRNA転写産物を濃縮します。これにより、シーケンスリソースを選択した転写産物に集中させることができ、不要で存在量の多い転写産物によるデータの隠蔽を防ぐことができます。
ターゲットエンリッチメントは、より高解像度のデータが必要なRNA-seqプロジェクトで幅広く活用できます。全エクソームシーケンスからlncRNAトランスクリプトーム解析まで、カスタムプローブパネルを設計することで、対象のRNA転写産物を効率的に濃縮することが可能です。
ターゲットRNA-seqアプローチの利点は、Morlionら(2021)のプレプリント論文で強調されています。この研究チームは、Twist Bioscienceが合成した565,878個のターゲットキャプチャプローブからなるカスタムパネルを活用しました。比較対象として、mRNAやlncRNAを含む複数のRNAの検出を目的とするトータルRNA-seqも使用されました。
研究では、がん研究者が遭遇する可能性のあるさまざまなサンプルタイプ(精液中の血漿、血小板除去血漿、FFPE腫瘍サンプル、MAQCA/B)を用いて比較した結果、以下の成果が示されました。
- FFPE、MAQCA/B、精液血漿サンプルにおいて、それぞれ3.5倍、4倍、8倍のリードがlncRNAにマッピングされる割合が増加
- ユニークに検出されるlncRNA転写産物が2倍に増加
- トータルRNA-seqでは検出されなかった数千種類のlncRNA転写産物の再現可能な同定
- トータルRNA-seqで見逃された5つのがん関連lncRNA転写産物の同定
- がん関連lncRNA転写産物のカバレッジが平均6倍に向上
これらの結果から、ターゲットRNA-seqはlncRNAの詳細な解析およびがん研究において強力なツールとなることが示されています。
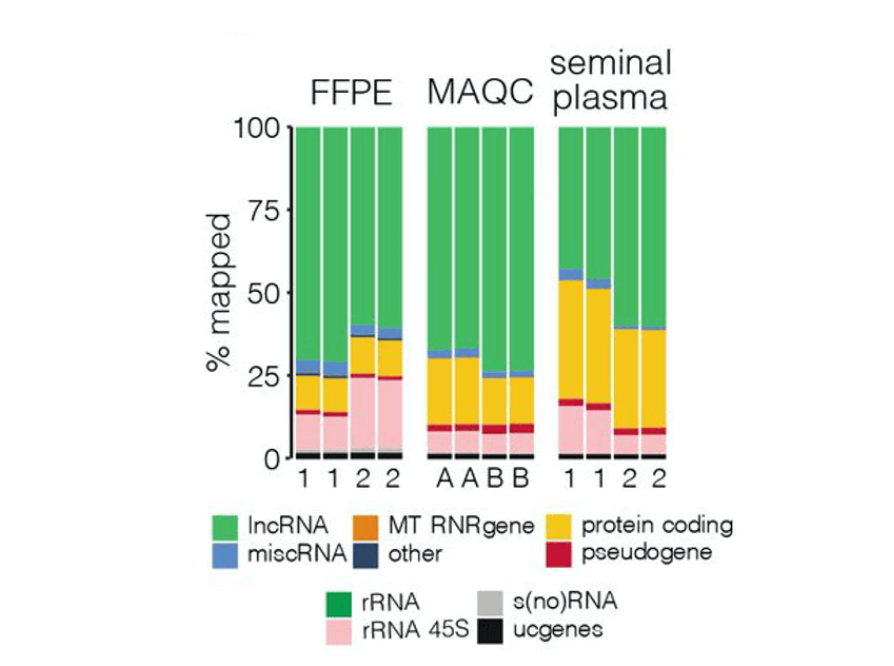
ターゲットキャプチャベースのRNAシーケンス(RNA-seq)は、lncRNAを検出するための高感度なツールです。データは、さまざまなサンプルタイプにおけるトータルRNA-seqのマッピングリードの割合を示しています。FFPEはホルマリン固定パラフィン包埋サンプル、MAQCは定量PCRヒト参照トータルRNAサンプルを指します。データはMorlionら(図1a)から引用され、CC-by-4.0ライセンスの下で利用可能です。
簡単に言えば、lncRNA転写産物を標的にして濃縮するために均一なプローブライブラリを設計することで、研究チームはトータルRNAシーケンスという従来のゴールドスタンダードと比較して、シーケンスのカバレッジ、感度、および再現性を大幅に向上させることに成功しました。
ターゲットRNA-seq向けカスタムターゲットエンリッチメントパネル
Morlionらの研究結果は、RNAシーケンスがターゲットエンリッチメントと組み合わせることでより高感度になるという証拠をさらに強化しています。しかし、この可能性を最大限に引き出すには、均一性とオンターゲット率の両方を最適化したプローブライブラリの慎重な設計が不可欠です。
オンターゲット率とは、キャプチャされた配列のうち目的のターゲットをどれだけ含んでいるかを示す指標です。均一性は、目的の転写産物がどれだけ均等にキャプチャされているかを示します。均一性とオンターゲット率を最適化することで、シーケンスの指標が改善され、より感度の高い実験が可能になります。これは、lncRNAのように検出が難しいヌクレオチドタイプを扱う場合に特に重要です。
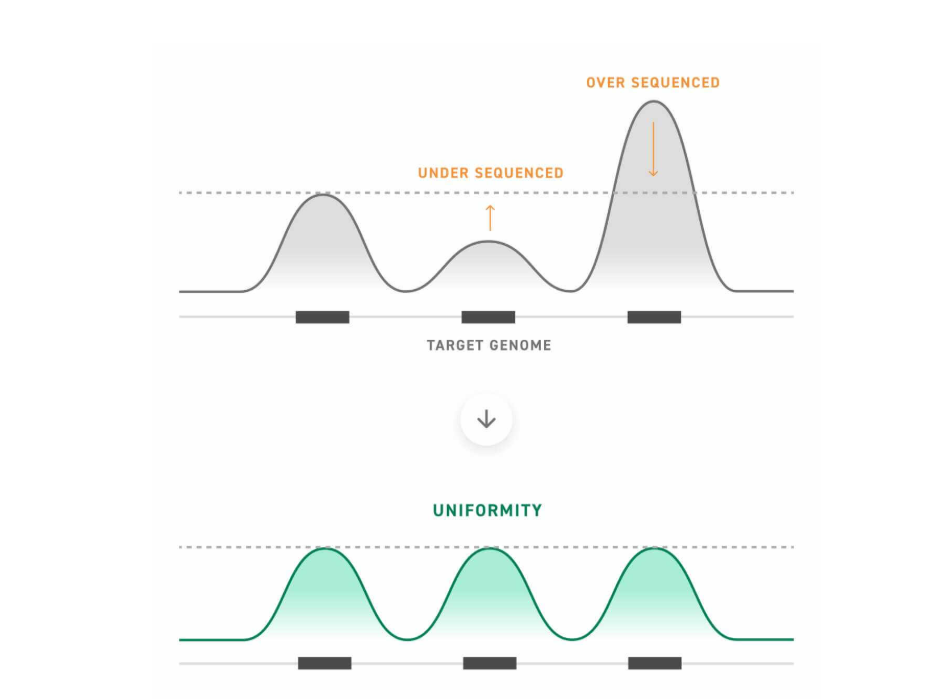
均一性とは、ゲノムの標的領域におけるリード分布を示す指標です。均一性が最適化されていないRNAターゲットエンリッチメントライブラリ(上部パネル参照)では、発現量の高いターゲットが過剰にシーケンスされ、発現量の低いターゲットが過小評価されてしまいます。一方、均一性を考慮して設計されたライブラリ(下部パネル参照)は、より均等で感度の高いカバレッジを提供します。
Twist Bioscienceでは、このような高品質なターゲットエンリッチメントパネルの設計と構築を専門としています。
この研究をサポートするため、私たちはカスタム設計されたターゲットエンリッチメントプローブパネルを提供しました。シリコンベースの高スループットDNA合成プラットフォームを活用することで、高い均一性を持つビオチン化二本鎖DNAプローブのパネルを迅速に製造し、実験に即座に活用できる形で提供しました。
ターゲットエンリッチメントとパネル均一性についてもっと知りたい方はこちらをご覧ください:
- ホワイトペーパー:効率的なターゲットNGSのためのカバレッジ均一性の重要性
- 15分ウェビナー:液体生検のRNAシーケンスにおけるカスタムエンリッチメント
lncRNAの臨床的意義
増え続ける証拠によると、lncRNA転写産物は基本的な細胞生物学においていくつかの機能を果たすことが示されています。例えば、転写および転写後の遺伝子制御や、タンパク質の局在を指示する役割が挙げられます(参考文献1)。また、lncRNAの中には腫瘍抑制因子として機能するものがある一方で、がんの発症や進行に深く関与しているものも存在します(参考文献4,6)。
lncRNAは、細胞特異的かつ時間依存的に発現するため、診断および治療法開発の両方に活用できる可能性があります(参考文献6)。このテーマに関する詳細なレビューも存在します。
lncRNAが臨床的に有意義なバイオマーカーとして確立されるためには、さらなる研究が必要です。研究者は、希少または低発現のlncRNAをさまざまな条件下で正確に同定し、定量化し、解析できるようになる必要があります。そのためには、ここで紹介したターゲットRNA-seqのような高感度で効率的なアッセイが不可欠です。
カスタムターゲットエンリッチメントパネルがどのように研究をサポートするかについてはこちらをご覧ください >
参考文献
- Public Health England. National Disease Registration Service: Case-mix adjusted percentage of cancers diagnosed at stages 1 and 2 in England, by Clinical Commissioning Group. May 2020. (Accessed 3rd June 2021).
- Yao, Run-Wen, et al. “Cellular Functions of Long Noncoding RNAs.” Nature Cell Biology, vol. 21, no. 5, 2019, pp. 542–551., doi:10.1038/s41556-019-0311-8.
- Bussotti, Giovanni et al. “Detecting and comparing non-coding RNAs in the high-throughput era.” International journal of molecular sciences vol. 14,8 15423-58. 24 Jul. 2013, doi:10.3390/ijms140815423
- Lorenzi, Lucía, et al. “Long Noncoding RNA Expression Profiling in Cancer: Challenges and Opportunities.” Genes, Chromosomes and Cancer, vol. 58, no. 4, 2019, pp. 191–199., doi:10.1002/gcc.22709.
- Djebali, Sarah et al. “Landscape of transcription in human cells.” Nature vol. 489,7414 (2012): 101-8. doi:10.1038/nature11233
- Shi, Ting et al. “Long Noncoding RNAs as Novel Biomarkers Have a Promising Future in Cancer Diagnostics.” Disease markers vol. 2016 (2016): 9085195. doi:10.1155/2016/9085195

